IL DNA CONTIENE IL CODICE DELLA VITA
La genetica è lo studio dei caratteri ereditari e di come si trasmettono le generazioni successive. I geni sono localizzati nei cromosomi i quali sono in grado di portare una quantità enorme di informazioni estremamente complesse,sono formati da atomi disposti in molecole.
Le prime analisi chimiche del materiale ereditario rivelarono che il cromosoma eucariote è costituito sia da acido desossiribonucleico (DNA) sia da proteine e che queste sostanze erano presenti nei cromosomi in quantità più o meno uguali, entrambi questi tipi di molecole avrebbero potuto avere il ruolo di materiale genetico. Le proteine sono molecole di amminoacidi presenti nelle cellule in 20 tipi diversi; il DNA è un polimero formato da 4 differenti tipi di nucleotidi. Gli amminoacidi potevano disporsi in moltissimi modi diversi, capaci di fornire le istruzioni per le numerose attività cellulari.
Il DNA venne isolato per al prima volta dal medico tedesco Friedrich Miescher nel 1869, nello stesso anno in cui Darwin pubblicava "L'origine delle specie" e Mendel i trattati sulla genetica. La sostanza isolata era bianca, zuccherina, leggermente acida e contenente fosforo e, poiché era stata trovata solamente nei nuclei delle cellule venne chiamata acido nucleico. Il nome venne poi modificato in acido desossiribonucleico per distinguere questa sostanza, in base alle sue caratteristiche chimiche da una simile, L'acido ribonucleico (RNA).
Il DNA è costituito da nucleotidi, il quale è a sua volta composto da una base azotata, da uno zucchero a 5 atomi di carbonio chiamato desossiribosio e da un gruppo fosfato. Vi sono due tipi di basi azotate: le purine che presentano una struttura simile a due anelli, e le pirimidine che hanno un solo anello. Nel DNA vi sono due tipi di purine: l'adenina e la guanina,e due tipi di pirimidine: la citosina e la timina. Nel 1943 il medico canadese Oswald T. Avery e i suoi collaboratori erano arrivati a stabilire che fosse il DNA il materiale genetico della cellula.
Intorno al 1940 ebbe inizio una serie di esperimenti fondamentali che utilizzavano un tipo di organismi particolarmente adatti per la sperimentazione, tali organismi erano i virus che aggrediscono i batteri (batteriofagi), quelli scelti per questi esperimenti furono Escherichia coli. L'analisi chimica dei batteriofagi rivelò che sono costituiti quasi esclusivamente da DNA e proteine. La questione su quale molecola costituisse i geni virali, cioè il materiale ereditario che dirige la sintesi dei nuovi virus all'interno della cellula batterica, fu risolto nel 1952 dagli scienziati Alfred D. Hershay e Martha Chase.
Le proteine contengono zolfo ma non fosforo, il DNA contiene fosforo ma non zolfo.
Hershey e Chase prepararono due campioni distinti di fagi: nel primo il DNA fagico venne marcato con l'isotopo radioattivo del fosforo 32P mentre l'altro presentava fagi con un involucro proteico contenente l'isotopo radioattivo dello zolfo 35S; i due isotopi radioattivi quindi marcavano rispettivamente, il DNA e le proteine.
Hersey e Chase coprirono che l'isotopo radioattivo 35S era rimasto fuori dalle cellule batteriche, insieme all'involucro virale vuoto, mentre l'isotopo radioattivo 32P era entrato nelle cellule ed era presente anche nelle nuove particelle virali. Grazie a questo esperimento s riuscì a dimostrare che solamente il DNA dei batteriofagi era coinvolto nel processo di riproduzione e che le proteine non potevano costituire il materiale ereditario.
Altre importanti scoperte furono:
il biologo molecolare statunitense Alfred Mirsky dimostrò che le cellule somatiche presenti in una qualsiasi specie di organismo contengono uguali quantità di DNA, mentre i gameti contengono esattamente la metà del DNA presente nelle cellule somatiche;
Erwin Chargaff analizzò il contenuto di purine e pirimidine del DNA di molte specie di essere viventi e giunse alla conclusione che le basi azotate non sono presenti n proporzioni uguali.
LA STRUTTURA DEL DNA
James Watson e Francis Crick erano interessati a studiare il DNA e ben presto cominciarono a lavorare insieme per cercare di risolvere il problema sulla sua struttura molecolare; essi non seguirono veri e propri esperimenti, ma condussero un esame razionale di tutti i dati allora conosciuti sul DNA, cercando di organizzarli in modo logico. Ai tempi in cui Watson e Crick iniziarono i loro studi erano già note parecchie informazioni: si sapeva che la molecola di DNA era di grandi dimensioni, lunga, filiforme ed era formata da nucleotidi, ognuno contenente una base azotata, una molecola di zucchero e un gruppo fosfato.
Nel 1950 Linus Pauling aveva dimostrato che le proteine possono avere una struttura elicoidale dovuta ai legami a idrogeno che i formano tra le spire adiacenti dell'elica. Pauling aveva anche avanzato l'ipotesi che la struttura del DNA potesse essere simile, ipotesi poi confermata dallo studio sui raggi X condotti da Maurice Wilkins e Rosalind Franklin. Infine vi erano i dati di Chargaff.
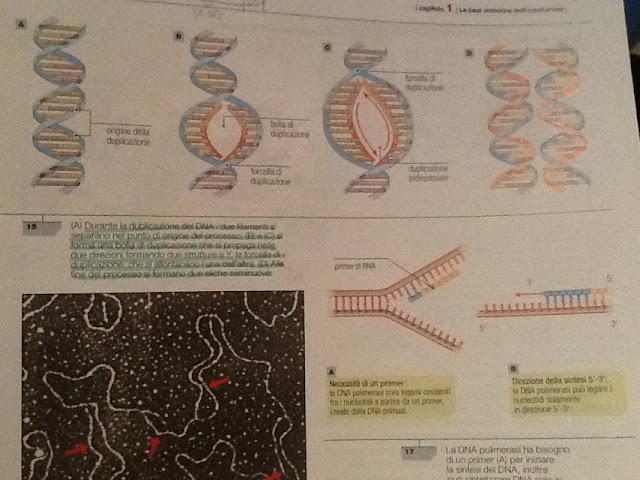
Watson e Crick cercarono di costruire un modello di DNA che fosse in accordo con con i fatti già noti e che spiegasse il suo ruolo biochimico. Le molecole di DNA dovevano essere eterogenee e varie, doveva esistere un qualche meccanismo di duplicazione rapido e preciso, capace di riprodurre copie fedeli del patrimonio genetico da trasmettere.
Watson e Crick furono in grado di dedurre che il DNA è una doppia elica molto lunga e spiralizzata.
Inoltre giunsero alla conclusione che una purina poteva legarsi solamente con una pirimidina, la combinazione di due purine raggiungerebbe più di 2 nanometri, mentre due pirimidine appaiate occuperebbero meno di 2 nanometri. Solo se una purina fosse appaiata con una pirimidina ci sarebbe stata una perfetta corrispondezza, e in ogni suo punto la molecola di DNA avrebbe presentato sempre la stessa lunghezza. I nucloetidi potevano essere disposti in qualunque ordine. Un'altra scoperta emozionante si ebbe quando Watson e Crick cominciarono a costruire il filamento corrispondente. Essi si trovarono di fronte ad un'altro importante vincolo: non solo le purine non potevano appaiarsi con altre purine, ma a causa della struttura della basi azotate anche il numero di legami era prestabilito.
L'adenina poteva appaiarsi soltanto con la timina mediante due legami a idrogeno (A=T) e la guanina soltanto con la citosina formando tre legami a idrogeno.
I filamenti hanno una direzione; in ogni filamento ciascun gruppo fosfato è preceduto ed è seguito da una molecola di zucchero. DA una parte il gruppo fosfato si lega al quinto atomo di carbonio della molecola dello zucchero (ossia in posizione 5'), mentre dall'altra si attacca al terzo atomo di carbonio del suo anello ( in posizione 3'). All'interno della doppia elica i due filamento corrono in direzioni opposte: se su ciascun filamento la direzione dall'estremità 5' alla 3' venisse indicata con una freccia, le due frecce avrebbero verso opposte. I filamenti vengono perciò definiti antiparalleli.
Da questo modello di DNA emerge dunque che, lungo una catena della doppia elica i nucleotidi possono essere disposti in un ordine qualunque, almeno dal punto di vista chimico, ma la loro sequenza determinerà l'ordine dei nucleotidi dell'altra catena dato che le basi azotate.
LA DUPLICAZIONE DEL DNA
Un'altra caratteristica fondamentale del materiale genetico è la capacità di ottenere copie esatte di se stesso; nella struttura doppia e complementare dell'elica di DNA è contenuto il meccanismo che permetto questa autoriproduzione.
Al momento della duplicazione dei cromosomi, la molecola si apre lungo la linea mediana e le basi appaiate si separano a livello dei legami a idrogeno. A mano a mano che i due filamenti si separano, essi fungono da stampo: ciascuno dirige la sintesi di un nuovo filamento complementare che avviene grazie all'utilizzo dei nucleotidi già presenti nella cellula. Ogni filamento forma una copia esatta di quello a cui era appaiato originariamente e vengono prodotte alla fine due copie identiche tra loro uguali alla molecola iniziale.
La duplicazione del DNA si svolge durante la fase S del ciclo cellulare ed è l'evento portante della duplicazione dei cromosomi; necessita di un gran numero di enzimi differenti, ognuno specifico nel catalizzare una particolare fase del processo. Inizia sempre da una specifica sequenza di nucleotidi, detta origine della duplicazione, e richiede particolari proteine di attivazione ed enzimi che, spezzando i legami a idrogeno che tengono unite le basi azotate complementari, siano in grado di aprire la doppia elica. Una volta che si sono separati i due filamenti , altre proteine si attaccano ai singoli filamenti per tenerli separati. IN questo modo si rende possibile la fase successiva, ossia l'effettiva sintesi del nuovo filamento, che è catalizzata da un gruppo di enzimi noto come DNA polimerasi.
La regione in cui avviene la sintesi è chiamata bolla di duplicazione, A entrambe le stremità della bolla la molecola forma una struttura a Y, nota come forcella di duplicazione; la duplicazione avviene in due direzioni opposte rispetto al punto di origine, perciò viene definita bidirezionale. Quando si è completata la sintesi dei nuovi filamenti di DNA le due catene a doppio filamento si separano in due nuove doppie eliche costituite ciascuna da un vecchio filamento e uno nuovo: per questo motivo al duplicazione viene definita semi-conservativa.
Quando la molecola di DNA comincia a duplicarsi, su ogni filamento in via di formazione, l'enzima DNA polimerasi comincia ad aggiungere i nuovi nucleotidi complementari a partire da una sequenza provvisoria chiamata innesco, o primer, che viene sintetizzata e inserita sul filamento stampo del DNA grazie all'enzima RNA primase; tale sequenza è di solito formata da una dozzina d nucleotidi di RNA ed è indispensabile perché l'enzima DNA polimerasi non riuscirebbe a entrare in azione e posizionare i nuovi nucleotidi.
Questi nucleotidi sono presenti nella cellula e posseggono inizialmente tre gruppi fosfato (desossiribonucleotidi trifosfato dNTP); nel momento dell'inserimento, l'enzima DNA polimerasi spezza il legame tra il primo e l secondo gruppo fosfato con il distacco di due gruppi fosfato, la rottura di questo legame covalente fornisce l'energia per unire i nucleotidi adiacenti. La duplicazione può avvenire soltanto in direzione 5'-3' dato che è l'unico gruppo fosfato rimasto del nuovo nucleotide può legarsi solo al carbonio del nucleotide precedente che si trovava in posizione 3'. Mentre il filamento complementare al filamento stampo che ha direzione 3'-5' può allungarsi in maniera continua senza interruzioni, l'altro filamento appaiandosi a un filamento a partire dalla sua estremità 5', dovrebbe allungarsi in direzione 3'-5'. Poiché ciò non è possibile, il filamento che ha come stampo il filamento con direzione da 5' a 3' viene assemblato al contrario.
Il filamento che può aggiungere nucleotidi senza interruzione, segue la forcella di duplicazione ed è detto filamento guida, mentre l'altro viene definito filamento in ritardo e viene sintetizzato in modo discontinuo sotto forma di singoli segmenti assemblati in direzione 5'-3' poi collegati tra loro, questi segmenti sono detti frammenti di Okazaki.
Sul filamento guida il punto di attacco, ossia il primer, per le DNA polimerasi è unico e si trova all'inizio del filamento da duplicare; sul filamento in ritardo i primer a cui le DNA polimerasi si devono attaccare sono molteplici e si trovano di volta in volta in prossimità della forcella di duplicazione.
Sul filamento in ritardo la DNA polimerasi non può catalizzare la sintesi dei frammenti di Okazaki se non trova il punto di attacco sul filamento da duplicare. A mano a mano che la doppia elica di DNA stampo si srotola, la DNA polimerasi consente la sintesi dei frammenti mancanti muovendosi correttamente in direzione 5'-3' e quindi in direzione opposta a quella in cui si muove al forcella di duplicazione.
Dopo essere state utilizzate,le sequenze innesco vengono eliminate e sostituite col DNA definitivo e i vari frammenti sono legati insieme dall'enzima DNA legasi in modo che anche questo filamento in via di formazione possa assumere un andamento continuo.
Diversi tipi di DNA polimerasi hanno anche la funzione di correggere alcuni errori che si potrebbero verificare durante questo processo; le DNA polimerasi sono in grado di aggiungere nucleotidi al filamento solo sono i nucleotidi già inseriti sono appaiati correttamente sul filamento stampo. Se si verifica un errore, questi enzimi invertono la propria direzione di marcia, rimuovendo ii nucleotidi fino a quando incontrano un nucleotide correttamente appaiato; a questo punto le DNA polimerasi iniziano di nuovo a muoversi in avanti aggiungendo i nucleotidi.
La capacità di leggere le sequenze e di rimuovere i nucleotidi è detta proofreading, essa garantisce una corretta duplicazione del DNA.
Ogni volta che incontrano una coppia di nucleotidi non correttamente appaiati, gli enzimi riparatori possono operare un taglio nel punto dell'errore e sostituire il nucleotide sbagliato con quello giusto; uno degli errori più comuni che può accadere è la perdita spontanea di una purina e la formazione di legami covalenti tra pirimidine adiacenti.
Nel 1986 il biochimico statunitense Kary B. Mullis mise a punto un metodo in laboratorio per produrre copie multiple dei frammenti di DNA. Mediante questa tecnica detta: reazione a catena della polimerasi (PCR), si è in grado di prendere da un piccolissimo campione di DNA e di sintetizzare in vitro milioni di copie di un suo specifico filamento.
Questa reazione a catena richiede la conoscenza delle sequenze di nucleotidi di ciascuna estremità del segmento di DNA che si vuole copiare. Il campione di DNA viene posto in una soluzione che contiene molecole di DNA-polimerasi. Il metodo si basa sulla ripetizione continua di tre reazioni successive:
I TELOMERI
Nei cromosomi eucarioti, le DNA polimerasi dei filamenti in ritardo non sono in grado di completare la sintesi dell'ultimo frammento di Okazaki in quanto ultimo primer; una volta rimosso, non può essere sostituito da DNA; alla sua prima duplicazione, quindi, ogni cromosoma tenderebbe ad accorciarsi perdendo così la sua normale funzionalità. Recentemente si è scoperto tuttavia che, a entrambe le estremità, molti cromosomi possiedono brevi sequenze nucleotidiche ripetute numerose volte, queste sequenze sono dette telomeri. A ogni duplicazione il filamento si accorcia per cui un cromosoma può duplicarsi anche 20-30 volte prima di perdere il suo contenuto genetico e far morire la cellula in cui si trova.
Questo è uno de motivi per cui le cellule vivono meno dell'organismo di cui fanno parte; tuttavia non tutte le cellule muoiono così in fretta; alcune hanno trovato il modo di risintetizzare il DNA telomerico perso durante la duplicazione e mantenere in tal modo integri i propri cromosomi. L'enzima che permette la ricostruzione dei telomeri mancanti si chiama telomerasi, si trova nel 90% delle cellule cancerose e consente loro di duplicarsi in modo continuo.
Gli scienziati hanno studiato l'enzima telomerasi anche in rapporto all'invecchiamento delle cellule; le cellule di vario tipo hanno una durata di vita piuttosto precisa che dipende dal numero di volte che esse si possono duplicare. Recenti esperimenti hanno dimostrato che in qualche caso è possibile, mediante l'inserimento nel DNA del gene che codifica per la telomerasi, allungare la vita delle cellule ritardando in questo modo il loro invecchiamento e, di conseguenza, l'invecchiamento del relativo tessuto corporeo.
Per maggiori informazioni : http://learn.genetics.utah.edu/content/begin/traits/telomeres/
Questa reazione a catena richiede la conoscenza delle sequenze di nucleotidi di ciascuna estremità del segmento di DNA che si vuole copiare. Il campione di DNA viene posto in una soluzione che contiene molecole di DNA-polimerasi. Il metodo si basa sulla ripetizione continua di tre reazioni successive:
- la soluzione viene scaldata in modo che i due filamenti della doppia elica di DNA si separino (denaturazione);
- la soluzione viene poi raffreddata per permettere ai primer di attaccarsi alla loro sequenza complementare (annealing);
- le molecole di DNA polimerasi, riconosciute le sequenze cominciano ad aggiungere nucleotidi a partire dai siti in cui sono attaccate tali sequenze.
I TELOMERI
Nei cromosomi eucarioti, le DNA polimerasi dei filamenti in ritardo non sono in grado di completare la sintesi dell'ultimo frammento di Okazaki in quanto ultimo primer; una volta rimosso, non può essere sostituito da DNA; alla sua prima duplicazione, quindi, ogni cromosoma tenderebbe ad accorciarsi perdendo così la sua normale funzionalità. Recentemente si è scoperto tuttavia che, a entrambe le estremità, molti cromosomi possiedono brevi sequenze nucleotidiche ripetute numerose volte, queste sequenze sono dette telomeri. A ogni duplicazione il filamento si accorcia per cui un cromosoma può duplicarsi anche 20-30 volte prima di perdere il suo contenuto genetico e far morire la cellula in cui si trova.
Questo è uno de motivi per cui le cellule vivono meno dell'organismo di cui fanno parte; tuttavia non tutte le cellule muoiono così in fretta; alcune hanno trovato il modo di risintetizzare il DNA telomerico perso durante la duplicazione e mantenere in tal modo integri i propri cromosomi. L'enzima che permette la ricostruzione dei telomeri mancanti si chiama telomerasi, si trova nel 90% delle cellule cancerose e consente loro di duplicarsi in modo continuo.
Gli scienziati hanno studiato l'enzima telomerasi anche in rapporto all'invecchiamento delle cellule; le cellule di vario tipo hanno una durata di vita piuttosto precisa che dipende dal numero di volte che esse si possono duplicare. Recenti esperimenti hanno dimostrato che in qualche caso è possibile, mediante l'inserimento nel DNA del gene che codifica per la telomerasi, allungare la vita delle cellule ritardando in questo modo il loro invecchiamento e, di conseguenza, l'invecchiamento del relativo tessuto corporeo.
Per maggiori informazioni : http://learn.genetics.utah.edu/content/begin/traits/telomeres/






.jpg)
Nessun commento:
Posta un commento